Desoxyribonukleinsäure
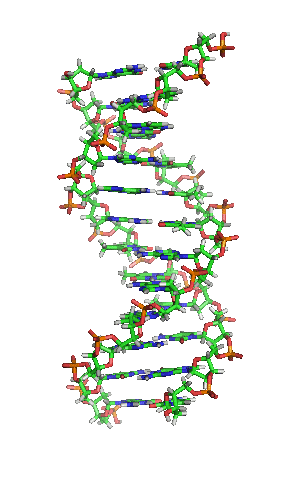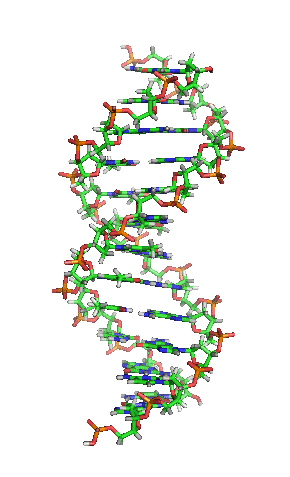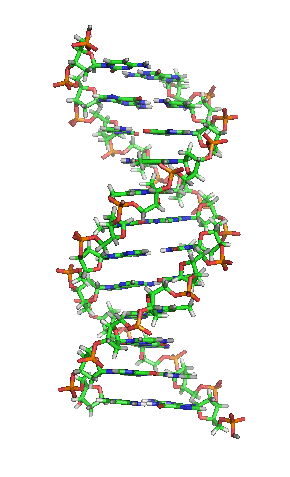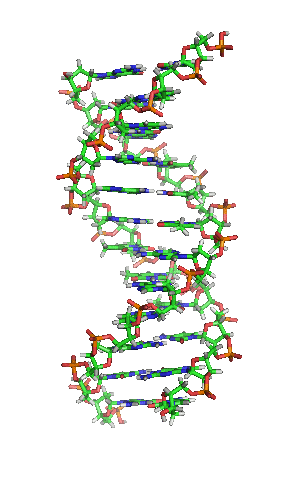
Desoxyribonukleinsäure (Des|oxy|ribo|nukle|in|säure; kurz DNS; englisch DNA für deoxyribonucleic acid) (lat.-fr.-gr. Kunstwort) ist ein in allen Lebewesen und DNA-Viren vorkommendes Biomolekül und Träger der Erbinformation, also der Gene.
Im Normalzustand ist DNA in Form einer Doppelhelix organisiert. Chemisch gesehen handelt es sich um Nukleinsäuren, lange Kettenmoleküle (Polymer) die aus vier verschiedenen Bausteinen, den Nukleotiden aufgebaut sind. Jedes Nukleotid besteht aus einem Phosphat-Rest, dem Zucker Desoxyribose und einer von vier organischen Basen (Adenin, Thymin, Guanin und Cytosin, oft abgekürzt mit A, T, G und C).
Die Gene in der DNA enthalten die Information für die Herstellung der Ribonukleinsäuren (RNA, im Deutschen auch RNS). Eine wichtige Gruppe von RNAs, die mRNAs enthält wiederum die Information für den Bau der Proteine (Eiweiße), welche für die biologische Entwicklung eines Lebewesens und den Stoffwechsel in der Zelle notwendig sind. Innerhalb der Protein-codierenden Gene legt die Abfolge der Basen die Abfolge der Aminosäuren des jeweiligen Proteins fest: Im genetischen Code stehen jeweils drei Basen für eine bestimmte Aminosäure.
In den Zellen von Eukaryoten, zu denen auch Pflanzen, Tiere und Pilze gehören, ist der Großteil der DNA im Zellkern als Chromosomen organisiert, ein kleiner Teil befindet sich in den Mitochondrien („Energiekraftwerken“ der Zelle). Pflanzen und Algen haben außerdem DNA in ihren Chloroplasten, den Photosynthese betreibenden Organellen. Bei Bakterien und Archaeen, den Prokaryoten, die keinen Zellkern besitzen, liegt die DNA im Cytoplasma. Manche Viren, die sogenannten RNA-Viren, haben keine DNA sondern RNA um die genetische Information zu speichern.
Im allgemeinen Sprachgebrauch wird die Desoxyribonukleinsäure überwiegend mit der englischen Abkürzung DNA bezeichnet; die parallel bestehende deutsche Abkürzung DNS wird hingegen seltener verwendet und ist laut Duden „veraltend“.[1]
Entdeckungsgeschichte



1869 entdeckte der Schweizer Arzt Friedrich Miescher in einem Extrakt aus Eiter eine aus den Zellkernen der Lymphozyten kommende Substanz, die er Nuklein nannte. Miescher arbeitete damals im Labor von Felix Hoppe-Seyler im Tübinger Schloss.[2] Erst 1919 identifizierte Phoebus Levene die Bestandteile der DNA (Base, Zucker und Phosphatrest).[3] Levene schlug eine kettenartige Struktur der DNA vor, in welcher die Nukleotide durch die Phosphatreste zusammengefügt sind und sich stetig wiederholen. 1937 publizierte William Astbury erstmals Röntgenbeugungsmuster, die auf eine repetitive Struktur der DNA hinwiesen.[4]
1943 wies Oswald Avery nach, dass die Transformation von Bakterien, das heißt die Weitergabe erblicher Information von einem Bakterien-Stamm auf einen anderen, auf der Übertragung von DNA beruht.[5] Dies widersprach der damals allgemein favorisierten Annahme, dass nicht die DNA, sondern Proteine die Träger der Erbinformation seien. Unterstützung in seiner Interpretation erhielt Avery 1952, als Alfred Hershey und Martha Chase nachwiesen, dass DNA die Erbinformation des T2-Phagen enthält.[6]
Der strukturelle Aufbau der DNA wurde erstmals 1953 von dem US-Amerikaner James Watson und dem Briten Francis Crick in ihrem berühmten Artikel Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid beschrieben[7]. Watson kam 1951 nach England, nachdem er ein Jahr zuvor an der Indiana University Bloomington in den USA promoviert hatte. Er hatte zwar ein Stipendium für Molekularbiologie bekommen, beschäftigte sich aber vermehrt mit der Frage des menschlichen Erbguts. Crick widmete sich in Cambridge gerade erfolglos seiner Promotion über die Kristallstruktur des Hämoglobinmoleküls, als er 1951 Watson traf.
Zu dieser Zeit war bereits ein erbitterter Wettlauf um die Struktur der DNA entbrannt, an dem sich neben anderen auch Linus Pauling am California Institute of Technology beteiligte. Watson und Crick waren eigentlich anderen Projekten zugeteilt worden und besaßen kein bedeutendes Fachwissen in Chemie. Sie bauten ihre Überlegungen auf den Forschungsergebnissen der anderen Wissenschaftler auf.
Watson sagte, er wolle das Erbgut entschlüsseln, ohne Chemie lernen zu müssen. In einem Gespräch mit dem renommierten Chemiker Erwin Chargaff vergaß Crick wichtige Molekülstrukturen und Watson machte im selben Gespräch unpassende Anmerkungen, die seine Unkenntnis auf dem Gebiet der Chemie verrieten. Chargaff nannte die jungen Kollegen im Anschluss „wissenschaftliche Clowns“.
Watson besuchte Ende 1952 am King's College in London Maurice Wilkins, der ihm DNA-Röntgenaufnahmen von Rosalind Franklin zeigte (was gegen den Willen von Franklin geschah). Watson sah sofort, dass es sich bei dem Molekül um eine Doppel-Helix handeln musste; Franklin selbst hatte aufgrund der Daten auch das Vorhandensein einer Helix vermutet, jedoch hatte sie kein überzeugendes Modell für die Struktur vorzuweisen. Da bekannt war, dass die Purin- und Pyrimidin-Basen Paare bilden, gelang es Watson und Crick, die ganze Molekularstruktur herzuleiten. So entwickelten sie am Cavendish-Laboratorium der Universität Cambridge das Doppelhelix-Modell der DNA mit den Basenpaaren in der Mitte, das am 25. April 1953 in der Zeitschrift Nature publiziert wurde.[8]
Diese denkwürdige Veröffentlichung enthält gegen Ende den Satz „It has not escaped our notice that the specific pairing we have postulated immediately suggests a possible copying mechanism for the genetic material“. (Es ist unserer Aufmerksamkeit nicht entgangen, dass die spezifische Paarung, die wir als gegeben voraussetzen, unmittelbar auf einen möglichen Vervielfältigungsmechanismus für das Erbgut schließen lässt.)
„Für ihre Entdeckungen über die Molekularstruktur der Nukleinsäuren und ihre Bedeutung für die Informationsübertragung in lebender Substanz“ erhielten Watson und Crick zusammen mit Maurice Wilkins 1962 den Nobelpreis für Medizin.[9]
Rosalind Franklin, deren Röntgenbeugungsdiagramme wesentlich zur Entschlüsselung der DNA-Struktur beigetragen hatten, war zu diesem Zeitpunkt bereits verstorben und konnte daher nicht mehr nominiert werden.
Für weitere geschichtliche Informationen zur Entschlüsselung der Vererbungsvorgänge siehe „Geschichte des Zellkerns“ sowie „Geschichte der Chromosomen“ und „Chromosomentheorie der Vererbung“.
Aufbau und Organisation
Bausteine
Die Desoxyribonukleinsäure ist ein langes Kettenmolekül (Polymer) aus vielen Bausteinen, die man Desoxyribonukleotide oder kurz Nukleotide nennt. Jedes Nukleotid hat drei Bestandteile: Phosphorsäure bzw. Phosphat, den Zucker Desoxyribose sowie eine heterozyklische Nukleobase oder kurz Base. Die Desoxyribose- und Phosphorsäure-Untereinheiten sind bei jedem Nukleotid gleich. Sie bilden das Rückgrat des Moleküls. Einheiten aus Base und Zucker (ohne Phosphat) werden als Nukleoside bezeichnet.
Die Phosphatreste sind aufgrund ihrer negativen Ladung hydrophil, sie geben DNA in wässriger Lösung insgesamt eine negative Ladung. Da diese negativ geladene, in Wasser gelöste DNA keine weiteren Protonen abgeben kann, handelt es sich streng genommen nicht (mehr) um eine Säure. Der Begriff Desoxyribonukleinsäure bezieht sich auf einen ungeladenen Zustand, in dem Protonen an die Phosphatreste angelagert sind.
Bei der Base kann es sich um ein Purin, nämlich Adenin (A) oder Guanin (G), oder um ein Pyrimidin, nämlich Thymin (T) oder Cytosin (C), handeln. Da sich die vier verschiedenen Nukleotide nur durch ihre Base unterscheiden, werden die Abkürzungen A, G, T und C auch für die entsprechenden Nukleotide verwendet.
Die fünf Kohlenstoffatome einer Desoxyribose sind von 1' (sprich Eins Strich) bis 5' nummeriert. Am 1'-Ende dieses Zuckers ist die Base gebunden. Am 5'-Ende hängt der Phosphatrest. Genau genommen handelt es sich bei der Desoxyribose um die 2-Desoxyribose; der Name kommt daher, dass im Vergleich zu einem Ribose-Molekül eine alkoholische OH-Gruppe an der 2'-Position fehlt (bzw. durch ein Wasserstoff ersetzt wurde).
An der 3'-Position ist eine OH-Gruppe vorhanden, welche die Desoxyribose über eine sogenannte Phosphodiester-Bindung mit dem 5'-Kohlenstoffatom des Zuckers des nächsten Nukleotids verknüpft (siehe Abbildung). Dadurch besitzt jeder sogenannte Einzelstrang zwei verschiedene Enden: ein 5'- und ein 3'-Ende. DNA-Polymerasen, die in der belebten Welt die Synthese von DNA-Strängen durchführen, können neue Nukleotide nur an die OH-Gruppe am 3'-Ende anfügen, nicht aber am 5'-Ende. Der Einzelstrang wächst also immer von 5' nach 3' (siehe auch DNA-Replikation weiter unten). Dabei wird ein Nukleosidtriphosphat (mit drei Phosphatresten) als neuer Baustein angeliefert, von dem zwei Phosphate in Form von Pyrophosphat abgespalten werden. Der verbleibende Phosphatrest des jeweils neu hinzukommenden Nukleotids wird mit der OH-Gruppe am 3'-Ende des letzten im Strang vorhandenen Nukleotids unter Wasserabspaltung verbunden. Die Abfolge der Basen im Strang codiert die genetische Information.
Die Doppelhelix

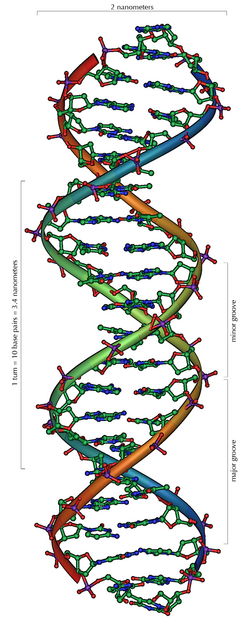

DNA kommt normalerweise als schraubenförmige Doppelhelix in einer Konformation vor, die „B-DNA“ genannt wird. Zwei der oben beschriebenen Einzelstränge sind dabei aneinandergelagert, und zwar in entgegengesetzter Richtung: An jedem Ende der Doppelhelix hat einer der beiden Einzelstränge sein 3'-Ende, der andere sein 5'-Ende. Durch die Aneinanderlagerung stehen sich in der Mitte der Doppelhelix immer zwei bestimmte Basen gegenüber, sie sind „gepaart“. Die Doppelhelix wird hauptsächlich durch Stapelwechselwirkungen zwischen aufeinander folgenden Basen stabilisiert (und nicht, wie oft behauptet, durch Wasserstoffbrücken).
Es paaren sich immer Adenin und Thymin, die dabei zwei Wasserstoffbrücken ausbilden, oder Cytosin mit Guanin, die über drei Wasserstoffbrücken miteinander verbunden sind. Eine Brückenbildung erfolgt zwischen den Molekülpositionen 1=1 sowie 6=6, bei Guanin-Cytosin-Paarungen zusätzlich zwischen 2=2. Da sich immer die gleichen Basen paaren, lässt sich aus der Sequenz der Basen in einem Strang die des anderen Strangs ableiten, die Sequenzen sind komplementär (siehe auch: Basenpaar). Dabei sind die Wasserstoffbrücken fast ausschließlich für die Spezifität der Paarung verantwortlich, nicht aber für die Stabilität der Doppelhelix.
Da stets ein Purin mit einem Pyrimidin kombiniert wird, ist der Abstand zwischen den Strängen überall gleich, es entsteht eine regelmäßige Struktur. Die ganze Helix hat einen Durchmesser von ungefähr 2 Nanometern (nm) und windet sich mit jedem Zuckermolekül um 0,34 nm weiter.
Die Ebenen der Zuckermoleküle stehen in einem Winkel von 36° zueinander, und eine vollständige Drehung wird folglich nach 10 Basen (360°) und 3,4 nm erreicht. DNA-Moleküle können sehr groß werden. Beispielsweise enthält das größte menschliche Chromosom 247 Millionen Basenpaare.[10]
Beim Umeinanderwinden der beiden Einzelstränge verbleiben seitliche Lücken, so dass hier die Basen direkt an der Oberfläche liegen. Von diesen Furchen gibt es zwei, die sich um die Doppelhelix herumwinden (siehe Abbildungen und Animation am Artikelanfang). Die „große Furche“ ist 2,2 nm breit, die „kleine Furche“ nur 1,2 nm.[11]
Entsprechend sind die Basen in der großen Furche besser zugänglich. Proteine, die sequenzspezifisch an die DNA binden, wie zum Beispiel Transkriptionsfaktoren, binden daher meist an der großen Furche[12].
Auch manche DNA-Farbstoffe, wie zum Beispiel DAPI, lagern sich an einer Furche an.
Die kumulierte Bindungsenergie zwischen den beiden Einzelsträngen hält diese zusammen. Kovalente Bindungen sind hier nicht vorhanden, die DNA-Doppelhelix besteht also nicht aus einem Molekül, sondern aus zweien. Dadurch können die beiden Stränge in biologischen Prozessen zeitweise getrennt werden.
Neben der eben beschriebenen B-DNA existieren auch eine „A-Form“ sowie eine 1979 von Alexander Rich und seinen Kollegen am MIT erstmals auch untersuchte, linkshändige, sogenannte „Z-DNA“. Diese tritt besonders in G-C-reichen Abschnitten auf. Erst 2005 wurde über eine Kristallstruktur berichtet, welche Z-DNA direkt in einer Verbindung mit B-DNA zeigt und so Hinweise auf eine biologische Aktivität von Z-DNA liefert[13]. Die folgende Tabelle und die daneben stehende Abbildung zeigen die Unterschiede der drei Formen im direkten Vergleich.
| Strukturmerkmal | A-DNA | B-DNA | Z-DNA |
|---|---|---|---|
| Drehsinn der Helix | rechts | rechts | links |
| Durchmesser | ≈ 2,6 nm | ≈ 2,0 nm | ≈ 1,8 nm |
| Basenpaare pro helikale Windung | 11,6 | 10,0 | 12 (6 Dimere) |
| Helikale Windung je Basenpaar (twist) | 31° | 36° | 60° (pro Dimer) |
| Ganghöhe (Anstieg pro Windung) | 3,4 nm | 3,4 nm | 4,4 nm |
| Anstieg pro Base | 0,29 nm | 0,34 nm | 0,74 nm (pro Dimer) |
| Neigungswinkel der Basenpaare zur Achse | 20° | 6° | 7° |
| Große Furche | eng und tief | breit und tief | flach |
| Kleine Furche | breit und flach | eng und tief | eng und tief |
| Zuckerkonformation | C3'-endo | C2'-endo | Pyrimidinbasen: C2'-endo Purinbasen: C3'-endo |
| Glykosidische Bindung | anti | anti | Pyrimidinbasen: anti Purinbasen: syn |
Die Stapel der Basenpaare (base stackings) liegen nicht wie Bücher exakt parallel aufeinander, sondern bilden Keile, die die Helix in die eine oder andere Richtung neigen. Den größten Keil bilden Adenosine, die mit Thymidinen des anderen Stranges gepaart sind. Folglich bildet eine Serie von AT-Paaren einen Bogen in der Helix. Wenn solche Serien in kurzen Abständen aufeinander folgen, nimmt das DNA-Molekül eine gebogene bzw. eine gekrümmte Struktur an, welche stabil ist. Dies wird auch Sequenz-induzierte Beugung genannt, da die Beugung auch von Proteinen hervorgerufen werden kann (die sogenannte Protein-induzierte Beugung). Sequenzinduzierte Beugung findet man häufig an wichtigen Stellen im Genom.
Chromatin und Chromosomen

Organisiert ist die DNA in der eukaryotischen Zelle in Form von Chromatinfäden, genannt Chromosomen, die im Zellkern liegen. Ein einzelnes Chromosom enthält jeweils einen langen, kontinuierlichen DNA-Doppelstrang. Da ein solcher DNA-Faden mehrere Zentimeter lang sein kann, ein Zellkern aber nur wenige Mikrometer Durchmesser hat, muss die DNA zusätzlich komprimiert bzw. „gepackt“ werden. Dies geschieht bei Eukaryoten mit sogenannten Chromatinproteinen, von denen besonders die basischen Histone zu erwähnen sind. Sie bilden die Nukleosomen, um die die DNA auf der niedrigsten Verpackungsebene herumgewickelt wird. Während der Kernteilung (Mitose) wird jedes Chromosom zu einer kompakten Form kondensiert. Dadurch werden sie lichtmikroskopisch bereits bei geringer Vergrößerung sichtbar.
Bakterielle und virale DNA
In prokaryotischen Zellen liegt die doppelsträngige DNA in den bisher dokumentierten Fällen mehrheitlich nicht als lineare Stränge mit jeweils einem Anfang und einem Ende vor, sondern als zirkuläre Moleküle – jedes Molekül (d.h. jeder DNA-Strang) schließt sich mit seinem 3'- und seinem 5'-Ende zum Kreis. Diese zwei ringförmigen, geschlossenen DNA-Moleküle werden je nach Länge der Sequenz als Bakterienchromosom oder Plasmid bezeichnet. Sie befinden sich bei Bakterien auch nicht in einem Zellkern, sondern liegen frei im Plasma vor. Die Prokaryoten-DNA wird mit Hilfe von Enzymen (zum Beispiel Topoisomerasen und Gyrasen) zu einfachen „Supercoils“ aufgewickelt, die einer geringelten Telefonschnur ähneln. Indem die Helices noch um sich selbst gedreht werden, sinkt der Platzbedarf für die Erbinformation. In den Bakterien sorgen Topoisomerasen dafür, dass durch ständiges Schneiden und Wiederverknüpfen der DNA der verdrillte Doppelstrang an einer gewünschten Stelle entwunden wird (Voraussetzung für Transkription und Replikation). Viren enthalten je nach Typ als Erbinformation entweder DNA oder RNA. Sowohl bei den DNA- wie den RNA-Viren wird die Nukleinsäure durch eine Protein-Hülle geschützt.
Chemische und physikalische Eigenschaften der DNA-Doppelhelix
Die DNA ist bei neutralem pH-Wert ein negativ geladenes Molekül, wobei die negativen Ladungen auf den Phosphaten im Rückgrat der Stränge sitzen. Zwar sind zwei der drei sauren OH-Gruppen der Phosphate mit den jeweils benachbarten Desoxyribosen verestert, die dritte ist jedoch noch vorhanden und gibt bei neutralem pH-Wert ein Proton ab, was die negative Ladung bewirkt. Diese Eigenschaft macht man sich bei der Agarose-Gelelektrophorese zu Nutze, um verschiedene DNA-Stränge nach ihrer Länge aufzutrennen. Einige physikalische Eigenschaften wie die freie Energie und der Schmelzpunkt der DNA hängen direkt mit dem GC-Gehalt zusammen, sind also sequenzabhängig.
Stapelwechselwirkungen
Für die Stabilität der Doppelhelix sind hauptsächlich zwei Faktoren verantwortlich: die Basenpaarung zwischen komplementären Basen sowie Stapelwechselwirkungen (stacking interactions) zwischen aufeinanderfolgenden Basen.
Anders als zunächst angenommen,[7] ist der Energiegewinn durch Wasserstoffbrückenbindungen vernachlässigbar, da die Basen mit dem umgebenden Wasser ähnlich gute Wasserstoffbrückenbindungen eingehen können. Die Wasserstoffbrücken eines GC-Basenpaares tragen nur minimal zur Stabilität der Doppelhelix bei, während diejenigen eines AT-Basenpaares sogar destabilisierend wirken.[14] Stapelwechselwirkungen hingegen wirken nur in der Doppelhelix zwischen aufeinanderfolgenden Basenpaaren: Zwischen den aromatischen Ringsystemen der heterozyklischen Basen entsteht eine dipol-induzierte Dipol-Wechselwirkung, welche energetisch günstig ist. Somit ist die Bildung des ersten Basenpaares aufgrund des geringen Energiegewinnes und des -verlustes recht ungünstig, jedoch die Elongation (Verlängerung) der Helix ist energetisch günstig, da die Basenpaarstapelung unter Energiegewinn verläuft.[15]
Die Stapelwechselwirkungen sind jedoch sequenzabhängig und energetisch am günstigsten für gestapelte GC-GC, weniger günstig für gestapelte AT-AT. Die Unterschiede in den Stapelwechselwirkungen erklären hauptsächlich, warum GC-reiche DNA-Abschnitte thermodynamisch stabiler sind als AT-reiche, während Wasserstoffbrückenbildung eine untergeordnete Rolle spielt.[14]
Schmelzpunkt
Der Schmelzpunkt der DNA ist die Temperatur, bei der die Bindungskräfte zwischen den beiden Einzelsträngen überwunden werden und diese sich voneinander trennen. Dies wird auch als Denaturierung bezeichnet.
Solange die DNA in einem kooperativen Übergang denaturiert (der sich in einem enggefassten Temperaturbereich vollzieht), bezeichnet der Schmelzpunkt die Temperatur, bei der die Hälfte der Doppelstränge in Einzelstränge denaturiert ist. Von dieser Definition sind die korrekten Bezeichnungen „midpoint of transition temperature“ bzw. Mittelpunktstemperatur abgeleitet.
Der Schmelzpunkt hängt von der jeweiligen Basensequenz in der Helix ab. Er steigt, wenn in ihr mehr GC-Basenpaare liegen, da diese entropisch günstiger sind als AT-Basenpaare. Das liegt nicht so sehr an der unterschiedlichen Zahl der Wasserstoffbrücken, welche die beiden Paare ausbilden, sondern viel mehr an den unterschiedlichen Stapelwechselwirkungen (stacking interactions). Die stacking-Energie zweier Basenpaare ist viel kleiner, wenn eines der beiden Paare ein AT-Basenpaar ist. GC-Stapel dagegen sind energetisch günstiger und stabilisieren die Doppelhelix stärker. Das Verhältnis der GC-Basenpaare zur Gesamtzahl aller Basenpaare wird durch den GC-Gehalt angegeben.
Da einzelsträngige DNA UV-Licht etwa 40 % stärker absorbiert als doppelsträngige, lässt sich die Übergangstemperatur in einem Photometer gut bestimmen.
Wenn die Temperatur der Lösung unter Tm zurückfällt, können sich die Einzelstränge wieder aneinanderlagern. Dieser Vorgang heißt Renaturierung oder Hybridisierung. Das Wechselspiel von De- und Renaturierung wird bei vielen biotechnologischen Verfahren ausgenutzt, zum Beispiel bei der Polymerase-Kettenreaktion (PCR), bei Southern Blots und der In-situ-Hybridisierung.
Kreuzförmige DNA an Palindromen
Ein Palindrom ist eine Folge von Nukleotiden, bei denen sich die beiden komplementären Stränge jeweils von rechts genauso lesen lassen wie von links.
Unter natürlichen Bedingungen (bei hoher Drehspannung der DNA) oder künstlich im Reagenzglas kann sich diese lineare Helix als Kreuzform (cruciform) herausbilden, indem zwei Zweige entstehen, die aus dem linearen Doppelstrang herausragen. Die Zweige stellen jeweils für sich eine Helix dar, allerdings bleiben am Ende eines Zweiges mindestens drei Nukleotide ungepaart. Beim Übergang von der Kreuzform in die lineare Helix bleibt die Basenpaarung wegen der Biegungsfähigkeit des Phosphodiester-Zucker-Rückgrates erhalten.
Die spontane Zusammenlagerung von komplementären Basen zu sog. Stamm-Schleifen-Strukturen wird häufig auch bei Einzelstrang-DNA oder -RNA beobachtet.
Genetischer Informationsgehalt und Transkription

DNA-Moleküle spielen als Informationsträger und „Andockstelle“ eine wichtige Rolle für Enzyme, die für die Transkription zuständig sind. Weiterhin ist die Information bestimmter DNA-Abschnitte, wie sie etwa in operativen Einheiten wie dem Operon vorliegt, wichtig für Regulationsprozesse innerhalb der Zelle.
Bestimmte Abschnitte der DNA, die sogenannten Gene, codieren für genetische Informationen, die Aufbau und Organisation des Organismus beeinflussen. Gene enthalten „Baupläne“ für Proteine oder Moleküle, die bei der Proteinsynthese oder der Regulation des Stoffwechsels einer Zelle beteiligt sind. Die Reihenfolge der Basen bestimmt dabei die genetische Information. Diese Basensequenz kann mittels Sequenzierung zum Beispiel über die Sanger-Methode ermittelt werden.
Die Basenabfolge (Basensequenz) eines Genabschnitts der DNA wird zunächst durch die Transkription in die komplementäre Basensequenz eines sogenannten Ribonukleinsäure-Moleküls überschrieben (abgekürzt RNA). RNA enthält im Unterschied zu DNA den Zucker Ribose anstelle von Desoxyribose und die Base Uracil anstelle von Thymin, der Informationsgehalt ist aber derselbe. Für die Proteinsynthese werden sogenannte mRNAs verwendet, einsträngige RNA-Moleküle, die aus dem Zellkern ins Zytoplasma hinaustransportiert werden, wo die Proteinsynthese stattfindet (siehe Proteinbiosynthese).
Nach der sog. „Ein-Gen-Ein-Protein-Hypothese“ wird von einem codierenden Abschnitt auf der DNA die Sequenz jeweils eines Proteinmoleküls abgelesen. Es gibt aber Regionen der DNA, die durch Verwendung unterschiedlicher Leseraster bei der Transkription jeweils mehrere Proteine codieren. Außerdem können durch alternatives Spleißen (nachträgliches Schneiden der mRNA) verschiedene Isoformen eines Proteins hergestellt werden.
Neben der codierenden DNA (den Genen) gibt es nichtkodierende DNA, die etwa beim Menschen über 90 % der gesamten DNA einer Zelle ausmacht.
Die Speicherkapazität der DNA konnte bisher nicht technisch nachgebildet werden. 1 Gramm getrockneter DNA enthält den Informationsgehalt von 1 Billion Compact Discs (CD). Mit der Informationsdichte in einem Teelöffel getrockneter DNA könnte die aktuelle Weltbevölkerung etwa 350 Mal nachgebaut werden.[16]
DNA-Replikation
Die DNA kann sich nach dem sog. semikonservativen Prinzip mit Hilfe von Enzymen selbst verdoppeln (replizieren). Die doppelsträngige Helix wird durch das Enzym Helikase aufgetrennt. Die entstehenden Einzelstränge dienen als Matrize (Vorlage) für den jeweils zu synthetisierenden komplementären Gegenstrang, der sich an sie anlagert.
Die DNA-Synthese, d. h. die Bindung der zu verknüpfenden Nukleotide, wird durch Enzyme aus der Gruppe der DNA-Polymerasen vollzogen. Ein zu verknüpfendes Nukleotid muss in der Triphosphat-Verbindung – also als Desoxyribonukleosidtriphosphat – vorliegen. Durch Abspaltung zweier Phosphatteile wird die für den Bindungsvorgang benötigte Energie frei.
Das Enzym Helikase bildet eine Replikationsgabel, zwei auseinander laufende DNA-Einzelstränge. In ihrem Bereich markiert ein RNA-Primer, der durch das Enzym Primase synthetisiert wird, den Startpunkt der DNA-Neusynthese. An dieses RNA-Molekül hängt die DNA-Polymerase nacheinander Nukleotide, die denen der DNA-Einzelstränge komplementär sind.
Die Verknüpfung der neuen Nukleotide zu einem komplementären DNA-Einzelstrang kann an den beiden alten Strängen nur in 5'→3' Richtung verlaufen und tut das demzufolge ohne Unterbrechung den alten 3'→5'-Strang entlang in Richtung der sich immer weiter öffnenden Replikationsgabel.
Die Synthese des neuen Stranges am alten 5'→3'-Strang dagegen kann nicht kontinuierlich auf die Replikationsgabel zu, sondern nur von dieser weg ebenfalls in 5'→3'-Richtung erfolgen. Der alte Doppelstrang ist aber zu Beginn der Replikation nur ein Stück weit geöffnet, so dass an dem zweiten Strang – in „unpassender“ Gegenrichtung – immer nur ein kurzes Stück neuer komplementärer DNA entstehen kann.
Da dabei eine DNA-Polymerase jeweils nur etwa 1000 Nukleotide verknüpft, ist es nötig, den gesamten komplementären Strang in einzelnen Stücken zu synthetisieren. Wenn sich die Replikationsgabel etwas weiter geöffnet hat, lagert sich daher ein neuer RNA-Primer wieder direkt an der Gabelungsstelle an den zweiten Einzelstrang an und initiiert die nächste DNA-Polymerase.
Bei der Synthese des 3'→5'-Stranges wird deshalb pro DNA-Syntheseeinheit jeweils ein neuer RNA-Primer benötigt. Primer und zugehörige Syntheseeinheit bezeichnet man als Okazaki-Fragment. Die für den Replikations-Start benötigten RNA-Primer werden anschließend enzymatisch abgebaut. Dadurch entstehen Lücken im neuen DNA-Strang, die durch spezielle DNA-Polymerasen mit DNA-Nukleotiden aufgefüllt werden.
Zum Abschluss verknüpft das Enzym Ligase die noch nicht miteinander verbundenen neuen DNA-Abschnitte zu einem einzigen Strang.
Mutationen und andere DNA-Schäden
Mutationen von DNA-Abschnitten – zum Beispiel Austausch von Basen gegen andere oder Änderungen in der Basensequenz – führen zu Veränderungen des Erbgutes, die zum Teil tödlich (letal) für den betroffenen Organismus sein können.
In seltenen Fällen sind solche Mutationen aber auch von Vorteil; sie bilden dann den Ausgangspunkt für die Veränderung von Lebewesen im Rahmen der Evolution. Mittels der Rekombination bei der geschlechtlichen Fortpflanzung wird diese Veränderung der DNA sogar zu einem entscheidenden Faktor bei der Evolution: Die eukaryotische Zelle besitzt in der Regel mehrere Chromosomensätze, d. h., ein DNA-Doppelstrang liegt mindestens zwei Mal vor. Durch wechselseitigen Austausch von Teilen dieser DNA-Stränge, dem Crossing-over bei der Meiose, können so neue Eigenschaften entstehen.
DNA-Moleküle können durch verschiedene Einflüsse beschädigt werden. Ionisierende Strahlung, wie zum Beispiel UV- oder γ-Strahlung, Alkylierung sowie Oxidation können die DNA-Basen chemisch verändern oder zum Strangbruch führen. Diese chemischen Änderungen beeinträchtigen unter Umständen die Paarungseigenschaften der betroffenen Basen. Viele der Mutationen während der Replikation kommen so zustande.
Einige häufige DNA-Schäden sind:
- die Bildung von Uracil aus Cytosin unter spontanem Verlust einer Aminogruppe durch Hydrolyse: Uracil ist wie Thymin komplementär zu Adenin.
- Thymin-Thymin-Dimerschäden verursacht durch photochemische Reaktion zweier aufeinander folgender Thyminbasen im DNA-Strang durch UV-Strahlung, zum Beispiel aus Sonnenlicht. Diese Schäden sind wahrscheinlich eine wesentliche Ursache für die Entstehung von Hautkrebs.
- die Entstehung von 8-oxo-Guanin durch Oxidation von Guanin: 8-oxo-Guanin ist sowohl zu Cytosin als auch zu Adenin komplementär. Während der Replikation können beide Basen gegenüber 8-oxo-Guanin eingebaut werden.
Aufgrund ihrer mutagenen Eigenschaften und ihres häufigen Auftretens (Schätzungen belaufen sich auf 104-106 neue Schäden pro Zelle und Tag) müssen DNA-Schäden rechtzeitig aus dem Genom entfernt werden. Zellen verfügen dafür über ein effizientes DNA-Reparatursystem. Es beseitigt Schäden mit Hilfe folgender Strategien:
- Direkte Schadensreversion: Ein Enzym macht die chemische Änderung an der DNA-Base rückgängig.
- Basenexcisionsreparatur: Die fehlerhafte Base, zum Beispiel 8-oxo-Guanin, wird aus dem Genom ausgeschnitten. Die entstandene freie Stelle wird anhand der Information im Gegenstrang neu synthetisiert.
- Nukleotidexcisionsreparatur: Ein größerer Teilstrang, der den Schaden enthält, wird aus dem Genom ausgeschnitten. Dieser wird anhand der Information im Gegenstrang neu synthetisiert.
- Homologe Rekombination: Sind beide DNA-Stränge beschädigt, wird die genetische Information aus dem zweiten Chromosom des homologen Chromosomenpaars für die Reparatur verwendet.
- Replikation mit speziellen Polymerasen: DNA-Polymerase η kann zum Beispiel fehlerfrei über einen TT-Dimerschaden replizieren. Menschen, bei denen Polymerase η nicht oder nur eingeschränkt funktioniert, leiden häufig an Xeroderma Pigmentosum, einer Erbkrankheit, die zu extremer Sonnenlichtempfindlichkeit führt.
Literatur
- Chris R. Calladine unter anderem: DNA – Das Molekül und seine Funktionsweise. 3. Aufl., Spektrum Akademischer Verlag, Heidelberg 2005, ISBN 3-8274-1605-1
- Terence A. Brown: Moderne Genetik. 2. Aufl., Spektrum Akademischer Verlag, Heidelberg 1999, ISBN 3-8274-0306-5
- Ernst Peter Fischer: Am Anfang war die Doppelhelix. Ullstein, Berlin 2004, ISBN 3-548-36673-2
- Ernst Peter Fischer: Das Genom. Fischer, Frankfurt M. 2002, ISBN 3-596-15362-X
- James D. Watson: Die Doppelhelix. Rowohlt, Reinbek 1997, ISBN 3-499-60255-5
- James D. Watson: Gene, Girls und Gamov. Piper, München 2003, ISBN 3-492-04428-X
- James D. Watson: Am Anfang war die Doppelhelix. Ullstein, Berlin 2003, ISBN 3-550-07566-9
- James D. Watson, M. Gilman, J. Witkowski, M. Zoller: Rekombinierte DNA. 2. Aufl., Spektrum Akademischer Verlag, Heidelberg 1993, ISBN 3-86025-072-8
- Rolf Knippers: Molekulare Genetik. 9. Aufl., G.Thieme Verlag, Stuttgart 2006, ISBN 3-13-477009-1
- Tomas Lindahl: Instability and decay of the primary structure of DNA. In: Nature. Nr. 362, 1993, 709-715, ISSN 0028-0836
- W. Wayt Gibbs: Preziosen im DNA-Schrott. In: Spektrum der Wissenschaft. Nr. 2, 2004, 68–75, ISSN 0170-2971
- W. Wayt Gibbs: DNA ist nicht alles. In: Spektrum der Wissenschaft. Nr. 3, 2004, 68–75, ISSN 0170-2971
- Hans-Jürgen Quadbeck-Seeger: Die Struktur der DNA – ein Modell-Projekt. Chemie in unserer Zeit, 42 (2008), 292-294
Weblinks
- Video: DNA-Isolierung aus Tomaten
- DNA Interactive – Seite des Cold Spring Harbor Institute und des Howard Hughes Medical Institute (eine exzellente Einführung in die Thematik, engl.)
- DNA from the Beginning des Dolan DNA Learning Center
- „DNA from the Beginning“ (deutsch)
- Nukleinsäuren
- „Übersetzer“ zum Finden der codierten Aminosäure zum codierenden Basentriplett oder umgekehrt
- „Übersetzer“ eines ganzen DNA-Abschnittes in die codierten Aminosäuren
Einzelnachweise
- ↑ Duden. Die deutsche Rechtschreibung. 24. Aufl., Mannheim 2006.
- ↑ Hubert Mania: Ein Opfer der wissenschaftlichen Vorurteile seiner Zeit. Die DNS wurde bereits 1869 im Tübinger Renaissanceschloss entdeckt. Telepolis. 17. April 2004.
- ↑ Levene P: The structure of yeast nucleic acid. In: J Biol Chem. 40, Nr. 2, 1919, S. 415–24.
- ↑ Astbury W: Nucleic acid. In: Symp. SOC. Exp. Bbl. 1, Nr. 66, 1947.
- ↑ Avery O, MacLeod C, McCarty M: Studies on the chemical nature of the substance inducing transformation of pneumococcal types. Inductions of transformation by a desoxyribonucleic acid fraction isolated from pneumococcus type III. In: J Exp Med. 79, Nr. 2, 1944, S. 137–158.
- ↑ Hershey A, Chase M: Independent functions of viral protein and nucleic acid in growth of bacteriophage. In: J Gen Physiol. 36, Nr. 1, 1952, S. 39–56. PMID 12981234.
- ↑ 7,0 7,1 Watson, J.D. & Crick F.H. (1953): Molecular structure of nucleic acids. A structure for deoxyribose nucleic acid. In: Nature. Bd. 171, Nr. 4356, S. 737–738. PMID 13054692, http://www.nature.com/nature/dna50/watsoncrick.pdf
- ↑ Katharina Kramer: Dem Leben auf der Spur. GEO kompakt Nr. 7 (2006)
- ↑ Informationen der Nobelstiftung zur Preisverleihung
- ↑ www.ensembl.org, Homo sapiens: Datenbankstand von Februar 2009. (Website auf Englisch)
- ↑ Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson R: Crystal structure analysis of a complete turn of B-DNA. In: Nature. 287, Nr. 5784, 1980, S. 755–758. PMID 7432492.
- ↑ Pabo C, Sauer R: Protein-DNA recognition. In: Annu Rev Biochem. 53, S. 293–321. PMID 6236744.
- ↑ Ha SC, Lowenhaupt K, Rich A, Kim YG, Kim KK: Crystal structure of a junction between B-DNA and Z-DNA reveals two extruded bases. In: Nature. 437, 2005, S. 1183–1186. PMID 16237447
- ↑ 14,0 14,1 Peter Yakovchuk, Ekaterina Protozanova and Maxim D. Frank-Kamenetskii. Base-stacking and base-pairing contributions into thermal stability of the DNA double helix. Nucleic Acids Research 2006 34(2):564-574; doi:10.1093/nar/gkj454 PMID 16449200
- ↑ Steger, G., Hrsg. (2003). Bioinformatik: Methoden zur Vorhersage von RNA- und Proteinstrukturen. Birkhäuser Verlag, Basel, Boston, Berlin.
- ↑ Leonard M. Adelmann: „Rechnen mit der DNA“. Spectrum der Wissenschaft, November 1998, S.77
| Dieser Artikel wurde am 21. Mai 2007 in dieser Version in die Liste der exzellenten Artikel aufgenommen. |